À l’occasion d’Alphy/AIEM 2023 nous avons lancé un premier concours (beta) Evoluthon.
Les règles et le contexte sont présentés plus bas. En deux mot il fallait reconstruire un arbre évolutif le plus proche de l’original à partir de 40 séquences générées par Aevol selon cet arbre.
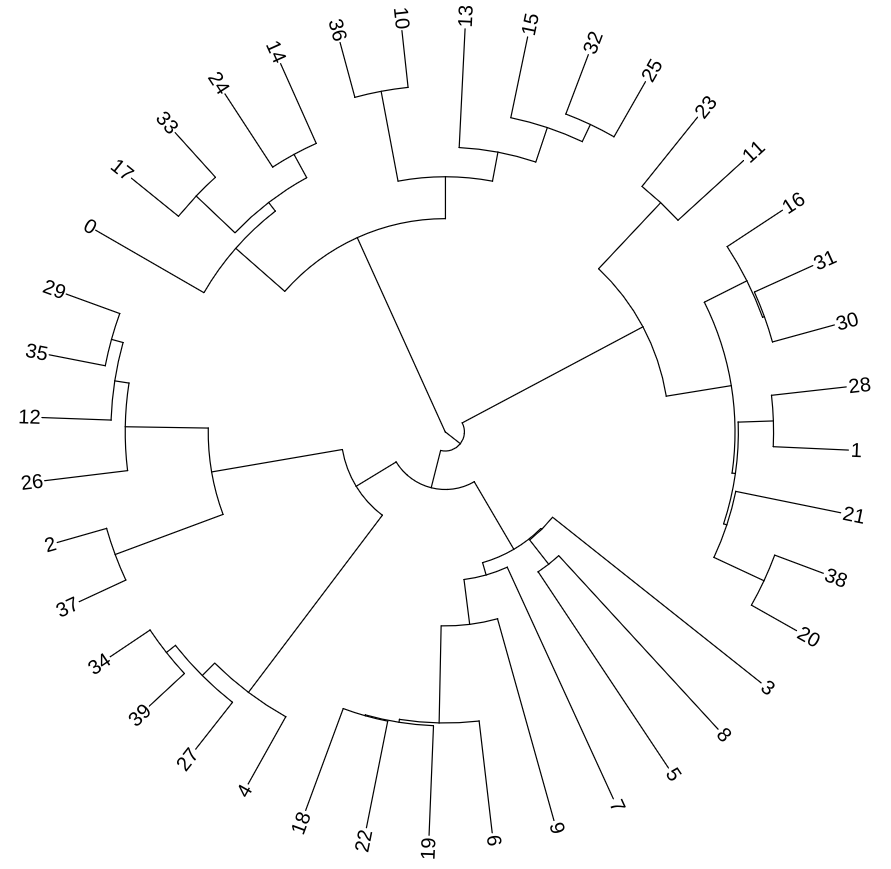
Maintenant que le concours est terminé, nous partageons l’arbre originel pour que tout le monde puisse s’entraîner sur ces premières séquences.
Nous lancerons un concours de plus grande envergure vers la rentrée avec plusieurs sets de séquences.
Ce nouveau concours devrait présenter différents niveaux de difficultés, avec des sets de séquences ayant évolué dans des condition différentes (bottleneck, changement de taux de mutation…).
Premier concours et solutions
Données
Pour télécharger les séquences en multi-fasta (les données de l’exercice), cliquez sur le lien suivant :
Solutions
Pour télécharger l’arbre originel (la « meilleure réponse/solution possible ») en format newick (en .txt ici), cliquez sur le premier lien ci-dessous.
Si vous voulez essayer de retrouver la séquence ancestrale, vous pouvez comparer à la vraie, téléchargeable via le lien suivant.
Si vous voulez essayer de déterminer les paramètres de la simulation (intitial population size, maximum indel size, point mutation rate, small deletion rate, small insertion rate, duplication rate, translocation rate, insertion rate, deletion rate), ils sont dévoilés dans le .txt du dernier lien.
Règles
Nous vous fournissons 40 séquences d’ADN d’environ 40 000 paires de bases simulées avec Aevol.
Elles sont inspirées de genome bactérien. Chaque séquence provient d’une espèce différente ; les espèces ont évolué in silico depuis un unique génome originel
utilisez vos méthodes et outils préférés pour reconstruire l’arbre phylogenetique le plus proche de l’originel.Vous pouvez soumettre vos arbres au format que vous voulez (newick, NEXUS, phyloxml…) en les envoyant à hugo.daudey@univ-lyon1.fr .
Aussi, n’hésitez pas à envoyer également toute sorte d’information que vous auriez trouvé sur l’évolution de ces séquences (tailles de pop., pression de sélection, événements notables…).
Nous pourrons vous confirmer ou infirmer vos déductions grâce à la collection des données que nous avons sur la simulation génératrice de ces séquences.
Contexte
Les méthodes d’évolution moléculaires font face à un problème de validation : on ne peux pas remonter dans le temps pour vérifier les hypothèses et prédictions concernant des événements qui se sont passé il y a des milliards d’années.
La méthode de validation la plus populaire dans la littérature est la simulation in silico.
L’évolution de génomes peut être simulée sur un nombre bien plus conséquent de générations qu’en évolution expérimentale, et de façon moins coûteuse
Les nouvelles méthodes sont presque toujours testées sur des simulations ad hoc, c’est-à-dire des simulations créées exprès pour tester spécifiquement ces nouvelles méthodes. Ce processus conduit inévitablement à intégrer des caractéristiques essentielles de la méthode testée dans les simulations, ce qui implique que ces simulations ne peuvent servir qu’à tester lesdites nouvelles méthodes, et généreront des instances ne représentant pas la complexité réelle des données.
Evoluthon est un projet qui vise à challenger des méthodes avec des données créées par des simulations non influencées par lesdites méthodes à tester. Pour y parvenir, nous proposons Aevol comme outil de benchmarking impartial.
Aevol est une plateforme de génétique numérique open source qui capture le processus évolutif à l’aide d’algorithmes génétiques et d’une modélisation au niveau de l’individu.
Les organismes numériques d’Aevol se reproduisent, rivalisent et mutent, évoluant pendant des centaines de milliers de générations sous une dynamique darwinienne typique.
Hugo Daudey (1), Marco Foley (2), Jonathan Rouzaud-Cornabas (2)(3), Vincent Daubin (1), Bastien Bousseau (1), Éric Tannier (1)(2), Guillaume Beslon (2)(3)
(1) Université Claude Bernard Lyon 1, CNRS, Laboratoire de Biométrie et Biologie Evolutive, Villeurbanne, France
(2) Centre de recherche Inria de Lyon (Équipe BEAGLE), Villeurbanne, France
(3) INSA-Lyon, Villeurbanne, France

Questions fréquentes:
- Pourquoi Aevol? Il est nécessaire que le logiciel de simulation soit développé par une équipe qui ne développe pas de méthode d’inférence, sinon la simulation pourrait ne pas être impartiale, mais biaisée vers un ou des modèle(s) particulier(s). Aevol est le seul logiciel de la sorte dont nous ayant connaissance.
- À quoi ces génomes sont ils censé ressembler ? Ils sont simulés avec un processus darwinien de mutation/sélection, et sont contraints par certaines limites en temps de calcul disponible. L’absence de sexe et leur taille les rapprochent des génomes bactériens. Cependant, de nombreuses caractéristiques peuvent ne pas ressembler à de vrais génomes bactériens. Imaginez-les comme une vie extraterrestre et voyez ce que vous pouvez déduire de leur évolution.



